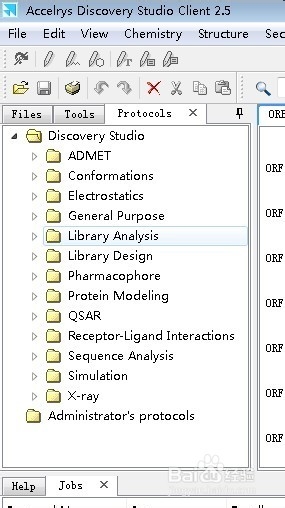
1、首先,确保电脑上已经成功安装Discovery Studio2.5软件(下面简称DS2.5),我有一篇经验写怎么安装DS。DS界面如下,主要是用左侧第三栏的protocol协议
2、首先,打开待模建的序列,DS对文本格式要求较高,fasta文件要求首行>蛋白名,下面另起一行是蛋白序列,如下图
3、打开待测序列文件,如下图
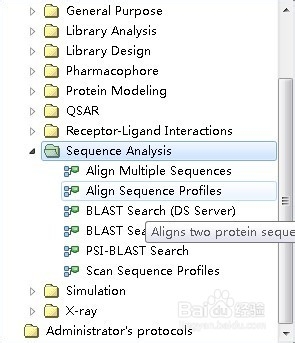
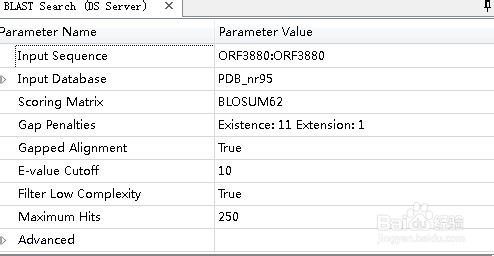
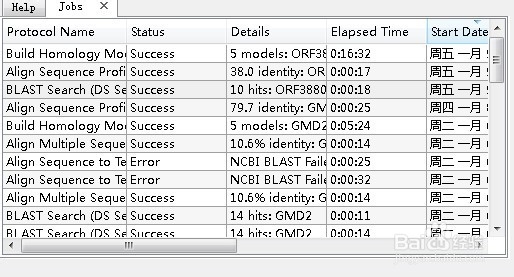
4、接下来,先做序列比对,搜索待测序列的模版,使用protocol的sequence analysis模块的blast search(DS sever),在下方打开功能模块,选项默认即可,点击窗口上方的绿色三角号,running!在左下方打开完成的job,列在最上方的模版是最佳模版,右键下载结构
5、在正式模拟前,需要将模版与模建序列比对,也是使用protocol的sequence analysis模块,点击align multiple sequences,type选项选择比对profiles,点击蓝箭头,running!比对结果,如下
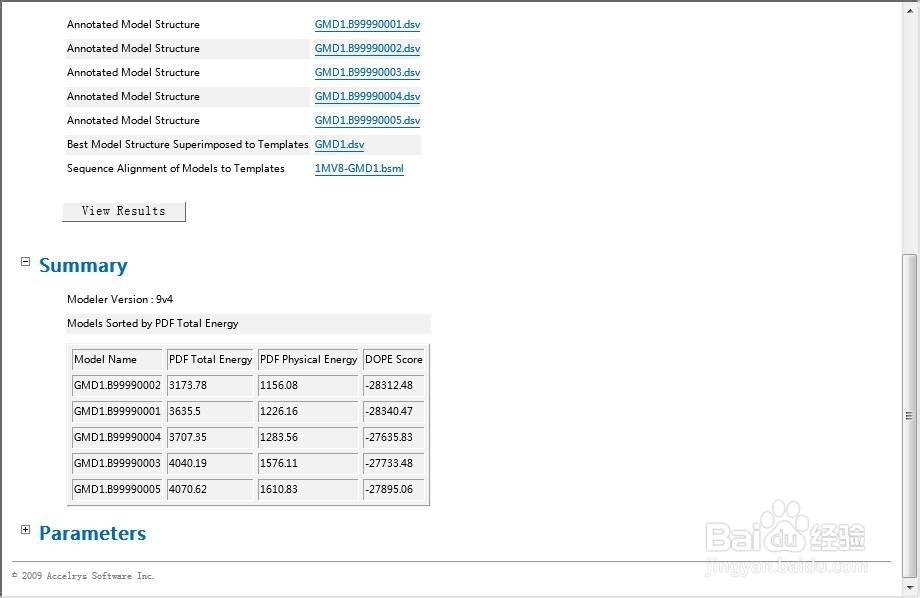
6、模拟,使用protein modeling模块的build homology models,输入第五步的序列比对结果,选择袷蜍滇刷模版和模建序列,点击蓝箭头,running!模建结果如下,根据dope score值选择最佳模建结果,一般值越小,结果越好。