1、数据准备:1、表型数据:第一列为基因型(品系)、第二列为环境、第三列为产量(观测值)2、图谱数据:第一行是标记名称,第一列是基因型(品种)名称,第二行和第三行是两个亲本的信息。3、基因型数据:第一列是标记名称,第二列是染色体,第三列是染色体上的位置。 没有行头。
2、打开GenStat软件
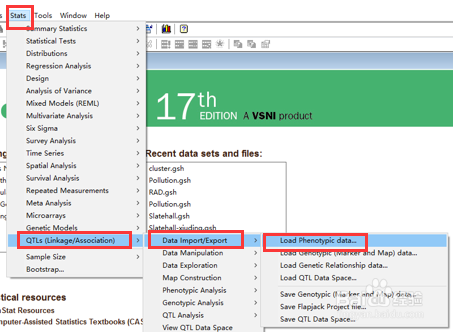
3、导入表型数据:•Stat->QTL(Linkage or Association)->data import->Loat phenotypic data1选择一个观测值yield导入到trait means2选择基因型G3选择环境E
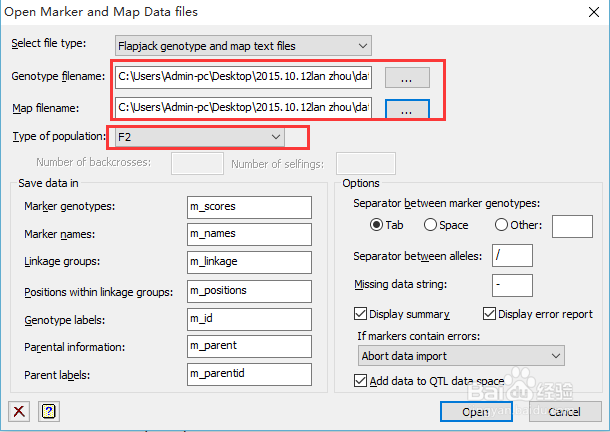
4、导入基因型数据和图谱数据:•Stat->QTL(Linkage or Association)->data import->load Genotypic (mark and map) data导入基因型数据导入表型数据选择群体来类型:F2
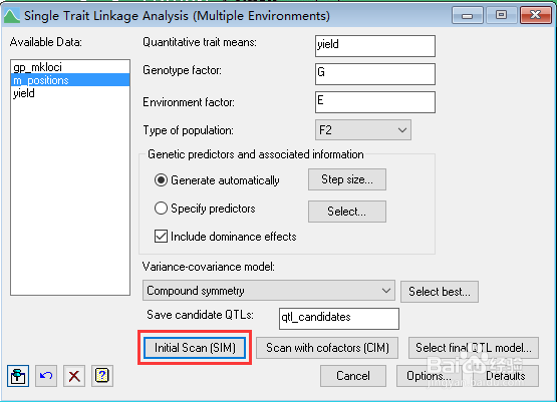
5、选择分析方法:单性状多环境连锁分析:QTL_analysis->Single Trait Linkage(Single Environment)
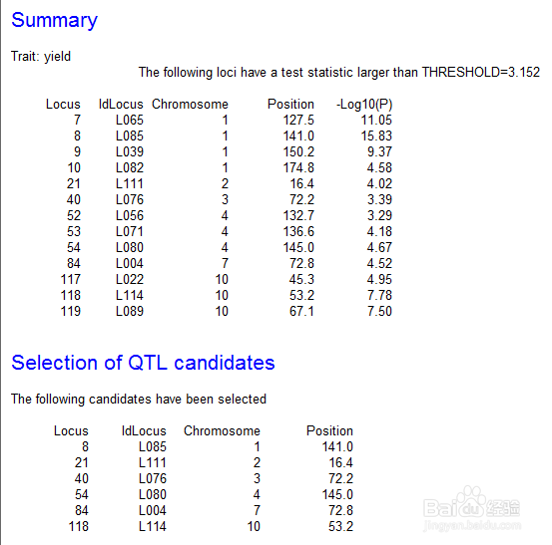
6、简单区间作图:SIM
7、SIM结果:
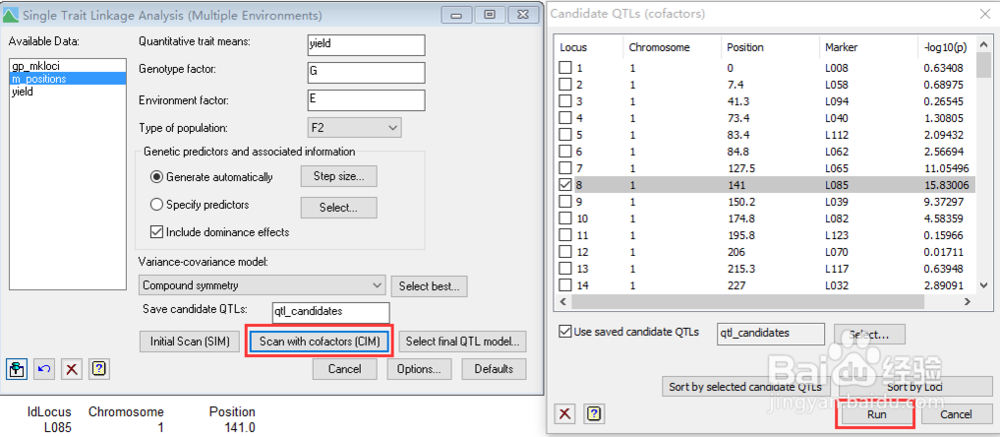
8、复合区间作图模型(CIM),默认辅助因子(cofactor),模型如下:
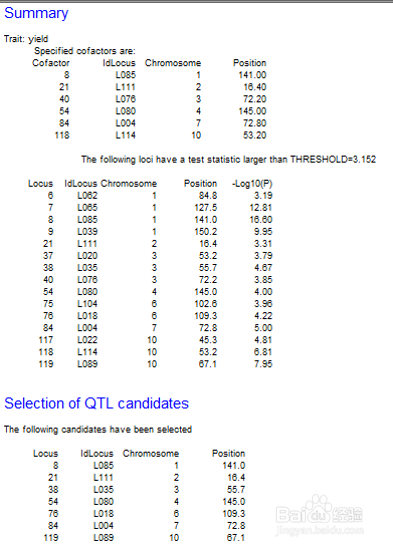
9、CIM结果:
10、QTL与环境互作模型:
11、结果一:用基于Reml的混合线性模型分析方法,查看方差分量,以及QTL X E的互作显著性检测
12、结果二:QTL X E的互作显著的QTL的信息,包括QTL的位点、效应、贡献率、贡献亲本
13、结果三:QTL在不同环境的贡献率
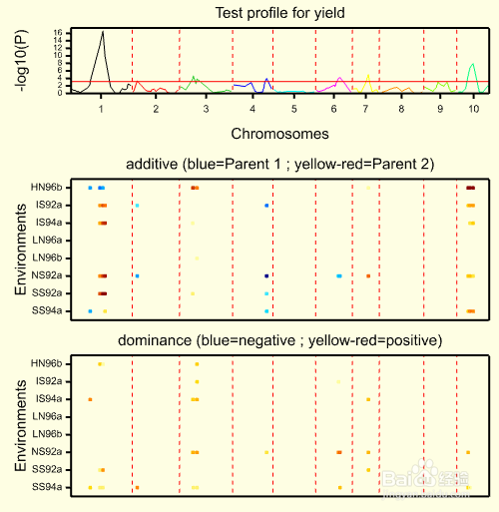
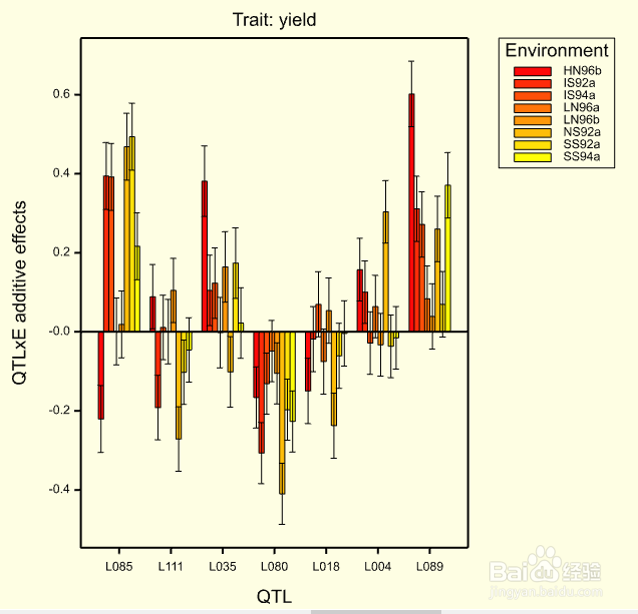
14、结果四:QTL与环境互作图形显示,图像显示共有7个QTL检测出与环境互作,这些QTL有的与环境互作正向,有的为负向,第七个QTL在所有环境中互作都为正向,考虑到这是产量的QTL定位,这个QTL可能更有意思。